Полимеразная цепная реакция (ПЦР)
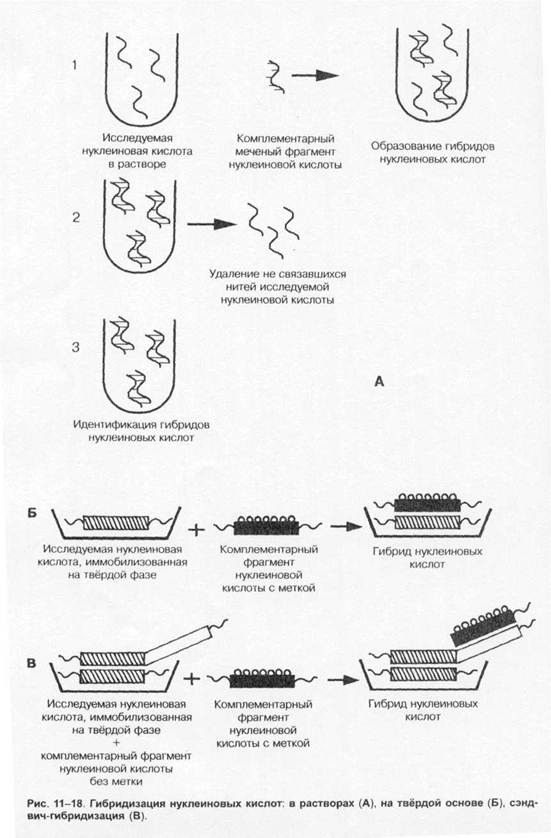
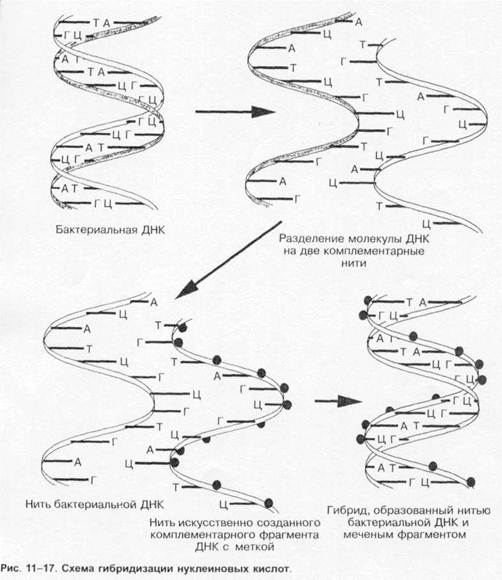
Основу метода ПЦР составляет катализируемое ДНК-полимеразой многократное образование копий определённого участка ДНК. Первоначально проводят отжиг — термическое разделение двухнитевой молекулы ДНК на отдельные цепочки. Затем среду охлаждают и вносят праймеры (затравки), комплементарные нуклеотидным последовательностям обеих цепочек. Для запуска реакции применяют синтетические праймеры — олигонуклеотиды, состоящие из 10-20 нуклеоти-дов (например, дезоксинуклеотидтрифосфат), взаимодействующие с окончаниями последовательностей и образующие последовательности в 50-1000 оснований. Затем в среду вносят термостабильную taq-полимеразу (по названию бактерии Thermus aquaticus), что запускает образование вторичных копий цепей ДНК, после чего образующиеся двухнитевые молекулы ДНК снова подогревают. Образующиеся отдельные цепочки остужают, вносят праймеры и снова повторяют процедуру подогрева и охлаждения; поскольку tag-полимераза термостабильна, то необходимость в её повторном внесении отсутствует (рис. 1-19).
ПЦР позволяет получить большие количества изучаемого фрагмента ДНК даже в том случае, если в распоряжении исследователя имеется всего лишь одна исходная молекула геномной ДНК.
Идентификацию копий ДНК
проводят методом электрофореза. Метод ПЦР лежит также в основе ДНК-идентификации личности, установления родства людей, выявления генов наследственных болезней и пр.
|
|
Микромир
В конце XIX — начале XX вв. физика вышла на уровень исследования микромира, для описания которого концептуальные построения классической физики оказались непригодными.
В результате научных открытий были опровергнуты представления об атомах как о последних неделимых структурных элементах материи.
Атомистическая гипотеза строения матери ...
Исследование способности штамма LPM-4 к ассимиляции
ЭДТА и глюкозы в процессе длительного культивирования с добавлением субстрата
В предыдущем разделе было показано, что ассимиляция глюкозы бактериальным штаммом LPM-4 индуцируется в процессе деградации ЭДТА, а кометаболизм ЭДТА и глюкозы у штамма LPM-4 не оказывает влияния на деградацию ЭДТА.
В данном разделе целью работы было исследование:
1) сохраняется ли способность клеток к деградации ЭДТА при дополнительно ...
Влияние психолептиков
на пептидергические системы. Нейропептиды при действии диазепама
Диазепам (7-хлор-1,3-дигидро-1-метил-5-фенил-2Н-1,4-бензодиазепин-2-он) является классическим транквилизатором, который проявляет анксиолитический, седативный, снотворный, миорелаксирующий, противосудорожный и другие эффекты [39]. Бензодиазепины были введены в клиническую практику около 40 лет назад и до сих пор находят широкое применен ...